
2023年3月7日�,來自俄羅斯科學院托木斯克國家醫(yī)學研究中心癌癥研究所癌癥進展生物學實驗室���、托木斯克國立大學轉(zhuǎn)化細胞與分子生物醫(yī)學實驗室����、俄羅斯人民友誼大學分子與細胞醫(yī)學研究所單細胞生物學實驗室等機構(gòu)的科學家們聯(lián)合在線發(fā)表了題為“Comparison of the Illumina NextSeq 2000 and GeneMind Genolab M sequencing platforms for spatial transcriptomics”的研究成果���。該研究基于真邁生物的GenoLab M和Illumina的NextSeq 2000兩個測序平臺分別對3例卵巢癌樣本進行10x?Genomics Visium空間轉(zhuǎn)錄組測序����,并對得到的測序數(shù)據(jù)進行比較分析���。研究發(fā)現(xiàn)GenoLab M在測序數(shù)據(jù)質(zhì)量方面的表現(xiàn)與NextSeq 2000不相伯仲��。在后續(xù)的降維分析���、細胞亞群分析及差異表達基因方面也與NextSeq 2000分析結(jié)果均保持了高度的一致性,證明其應(yīng)用于空間轉(zhuǎn)錄組的實力�。
背景介紹
單細胞轉(zhuǎn)錄組測序(scRNA-seq)是在單個細胞水平進行高通量轉(zhuǎn)錄組測序的一項新技術(shù),能夠有效解決細胞異質(zhì)性以及組織轉(zhuǎn)錄組測序(bulk RNA-seq)被掩蓋的細胞群內(nèi)轉(zhuǎn)錄組異質(zhì)性難題���。該技術(shù)有助于發(fā)現(xiàn)新的稀有細胞類型����,深入了解細胞生長與分化過程中的表達調(diào)控機制���。然而��,在進行scRNA-seq測序的細胞解離過程中不僅存在與細胞應(yīng)激�、細胞死亡/細胞聚集等相關(guān)的阻礙因素�,同時還會丟失空間信息。此外�,一些組織中的特定細胞類型,尤其是免疫細胞���,難以從組織中解離出來���,更限制了scRNA-seq測序的廣泛應(yīng)用。技術(shù)更迭��,空間轉(zhuǎn)錄組技術(shù)橫空出世��,更是在2020年被Nature Method評為年度技術(shù)�����。其中基于10X 單細胞平臺的空間轉(zhuǎn)錄組技術(shù)成為了主流。該技術(shù)不僅能夠檢測完整組織切片的總mRNA����,而且還能將組織的空間信息和形態(tài)學內(nèi)容與mRNA整合在一起,最終繪制出基因表達的空間位置信息���。為研究細胞功能����、細胞表型和組織微環(huán)境等提供了關(guān)鍵的信息�����。
真邁生物的GenoLab M測序儀作為一個新的測序平臺��,在轉(zhuǎn)錄組與lncRNA測序���、WGS和WES方面表現(xiàn)優(yōu)異�,是否可以應(yīng)用到空間轉(zhuǎn)錄組呢����?對此,研究使用3份卵巢癌樣本,分別在GenoLab M與NextSeq 2000上完成空間轉(zhuǎn)錄組測序����,比較兩個平臺測序結(jié)果的一致性。
結(jié)果概要
01實驗設(shè)計與測序質(zhì)量
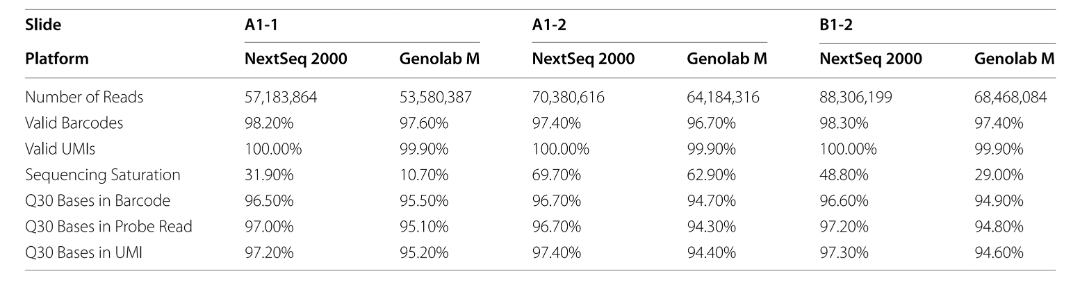 表1 三例卵巢癌樣本兩個測序平臺的測序質(zhì)量比較
表1 三例卵巢癌樣本兩個測序平臺的測序質(zhì)量比較
本研究將3份卵巢癌的石蠟切片樣本�����,分別構(gòu)建10xGenomics Visium的測序文庫��。隨后將3個文庫一分為二�,分別使用GenoLab M與NextSeq 2000測序平臺進行測序。2個平臺同一個樣本測序數(shù)據(jù)總量接近��,NextSeq 2000中有效barcode的百分比和有效UMI的百分比分別高出GenoLab M 0.74%和0.1%��。而Q30堿基的百分比在barcode����、探針和UMI對比中也比GenoLab M分別高出1.6%、2.2%和2.6%����。以上的差異主要是GenoLab M測序數(shù)據(jù)深度與測序reads duplication相對較低����。
* Barcode是含有空間位置標簽的一段核酸序列��,區(qū)分不同的spots�,每個spots包含數(shù)十個細胞用于定位;
* Probe探針是用于結(jié)合mRNA的的序列��;
* UMI是區(qū)分不同mRNA�,用于絕對定量的核酸序列;
02reads mapping與基因表達絕對定量
兩個平臺組織的spots下reads的比例基本一致����,差異不顯著。每個spots和組織的平均reads數(shù)與測序深度也基本一致����。除樣本B1-2外,GenoLab M平臺中每個spot的UMI計數(shù)和基因的中位數(shù)以及檢測到的基因數(shù)都比NextSeq 2000高�����。而NextSeq 2000在Reads Mapped Confidently to Probe Set 的表現(xiàn)中略高于GenoLab M��。這與探針reads更高的測序質(zhì)量一致。盡管如此���,在Reads Mapped Confidently to the Filtered Probe Set的對比中 GenoLab M仍然有比NextSeq 2000更好的表現(xiàn)����。
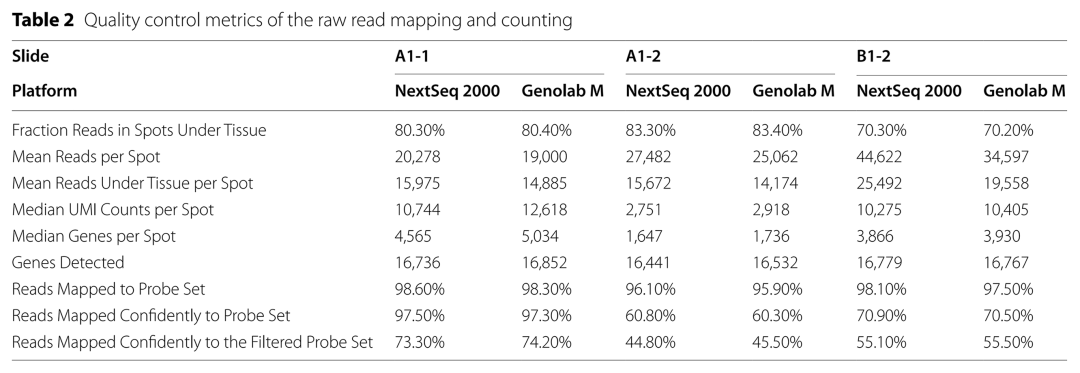 表2 reads mapping和UMI計數(shù)的測序質(zhì)量矩陣比較
表2 reads mapping和UMI計數(shù)的測序質(zhì)量矩陣比較
03基因��、UMIs�����、reads在組織的spots上的檢測情況
兩個測序平臺組織的spots下總的UMI和基因數(shù)目具有很高的一致性(Figure 1A)��。三個樣本的基因-UMI關(guān)系在兩個平臺的一致性也很高(Figure 1B)��。此外����,GC譜也高度一致(Figure 1C)���,說明兩個測序平臺都沒有明顯的測序偏向性�����。
接著研究者去除低表達基因和基因數(shù)目較少的spots后��,比較兩個平臺的基因交并集情況���。結(jié)果表明平臺特有基因相對于交集占比都比較小�,其中GenoLab M的特有基因稍多(Figure 1D)��。在兩個平臺上測序的spots基因交集之間存在很強的相關(guān)性�����。三個樣本A1-1����、A1-2 和 B1-2 樣本的平均相關(guān)系數(shù)分別為 0.82、0.90 和 0.99(Figure 1E)����。
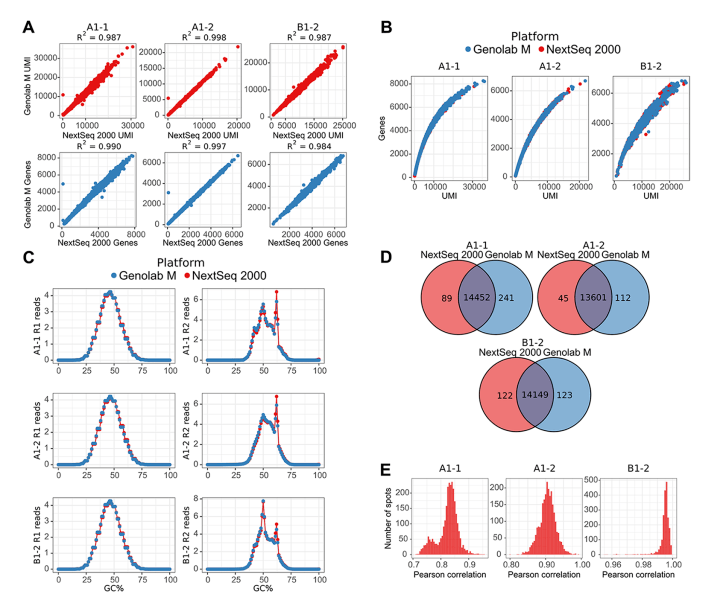 圖1 兩個測序平臺檢測到的reads數(shù)目����,UMI和基因的比較
圖1 兩個測序平臺檢測到的reads數(shù)目����,UMI和基因的比較
A 組織spots上轉(zhuǎn)錄本(基于UMI計數(shù))和基因數(shù)的一致性分析;
B 組織spots上基因的UMI分布�;
C reads的GC含量譜;
D 平臺間共有和特有基因的維恩圖���;
E SCT轉(zhuǎn)化基因交集的Pearson相關(guān)系數(shù)分布��;
04降維��、細胞亞群聚類和差異表達分析
三個樣本在兩個平臺的測序結(jié)果分析后得到的細胞亞群幾乎一致���,都是16個亞群(Figure 2A)���,來自不同測序平臺分析的樣本的組織spots所在的位置也非常接近(Figure 2B)����。A1-1含有7個亞群�����、A1-2有4個亞群、B1-2有5個亞群��。差異表達基因(DEG)檢測方面�,NextSeq 2000和GenoLab M分別檢測到的特有DEG約占所有檢測的DEG的16%;共有的占比更高為68%(Figure 2C-E)����。
* DEG的篩選條件,F(xiàn)DR<0.01且log fc=''>0.25(差異倍數(shù)>1.19)��;
* FDR False Discovery Rate錯誤發(fā)現(xiàn)率��;
* LTF Log2(Fold Change) 差異倍數(shù)fold change的log2值��;
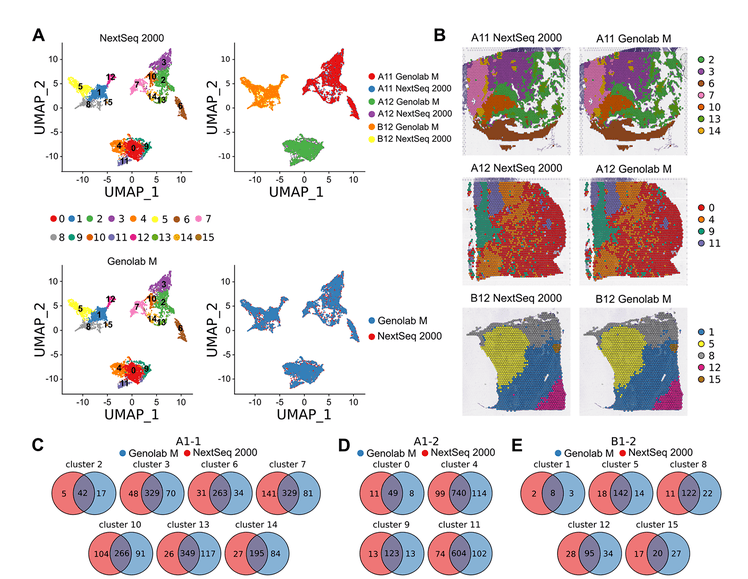 圖2 非線性降維���、聚類和差異表達分析
圖2 非線性降維���、聚類和差異表達分析
A 未做批次校正的兩個平臺的測序數(shù)據(jù)UMAP降維和聚類;
B 亞群的空間分布�����;
C A1-1樣本的平臺間共有與特有差異表達基因的維恩圖;
D A1-2樣本的平臺間共有與特有差異表達基因的維恩圖�;
E B1-2樣本的平臺間共有與特有差異表達基因的維恩圖;
05差異表達基因的深入分析
研究對平臺共有與特有的DEG深入分析以評估特有DEG的來源��。通過DEG的計數(shù)來可視化LFC和FDR的分布���。圖3A展示了DEG-LFC在組織spots亞群聚類的分布�,發(fā)現(xiàn)所有平臺特有的DEG的LFC均分布在所選閾值的邊緣����。圖3B展示了DEG-FDR在組織spots亞群聚類的分布,平臺特有DEG的FDR值往往最大(代表顯著性值最低)��。綜合來說�,平臺特有DEG比較接近閾值,而共有的DEG則具有較小的FDR和較高的差異倍數(shù)���。
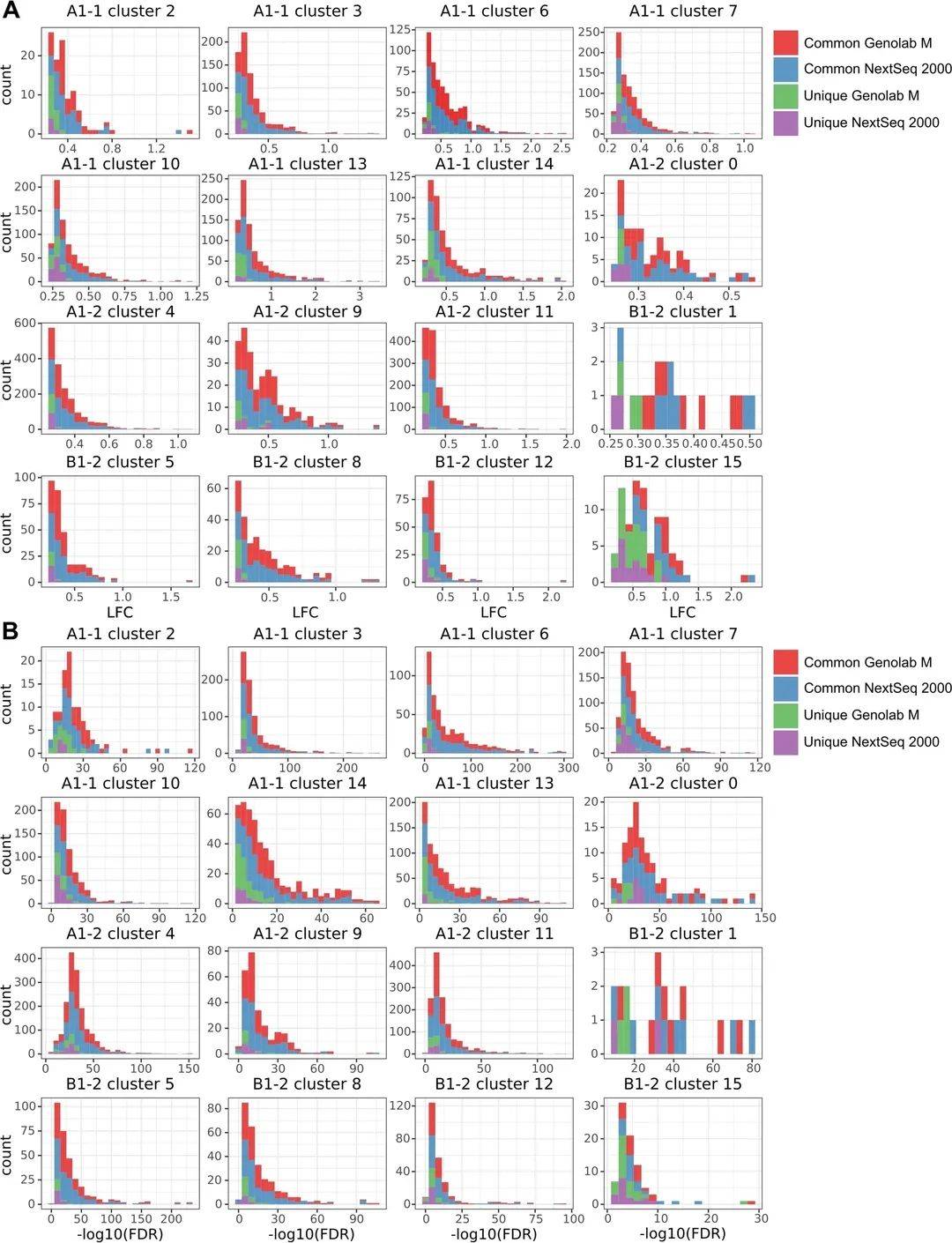 圖3 亞群差異表達的差異倍數(shù)(A)與FDR(B)的分布情況
圖3 亞群差異表達的差異倍數(shù)(A)與FDR(B)的分布情況
結(jié)論
研究首次比較了GenoLab M和NextSeq 2000在空間轉(zhuǎn)錄組測序的性能,兩個平臺均采用了可逆終止的邊合成邊測序的技術(shù)�。三份Visium的測序文庫在兩個測序平臺上得到了一致的空間轉(zhuǎn)錄組分析數(shù)據(jù)�����。研究結(jié)果表明GenoLab M的測序性能與NextSeq 2000接近��,適用于進行基于10?×?Genomics的空間轉(zhuǎn)錄組測序�。