近日���,真邁生物用戶俄羅斯莫斯科國立醫(yī)科大學�����、內分泌學研究中心等機構在cancers上發(fā)表了題為“A New Approach of Detecting?ALK?Fusion Oncogenes by RNA?Sequencing Exon Coverage Analysis”的科研成果,基于真邁生物的FASTASeq 300測序平臺��,開發(fā)一種新的生物信息學方法���,利用RNA測序數據中的外顯子覆蓋度分析來準確地預測臨床上顯著的ALK原癌基因融合�。在一小部分ALK陽性腫瘤中實現了96%的準確率(100%的靈敏度��,94.9%的特異性)。
ALK(間變性淋巴瘤激酶)基因重排在多種腫瘤類型中是驅動突變的常見來源�����。常用的檢測ALK融合的方法包括免疫組化(IHC)��、熒光原位雜交(FISH)���、反轉錄定量聚合酶鏈反應(RT-qPCR)和panel測序�。此外�����,全轉錄組測序能夠分析廣泛的癌癥生物標志物�����,包括失調基因��,分子途徑及腫瘤驅動基因的基因融合轉錄本�����。然而�,由于在融合斷點處覆蓋不足�����,難以發(fā)現融合位點����?����;诖?,本文開發(fā)了一種新的生物信息學方法���,可以從RNA測序數據中準確地預測ALK融合����。
丨ALK覆蓋不對稱篩選
文章通過使用TruSight和OncoFu兩個panel對樣本進行RNA建庫��,并使用FASTASeq 300對文庫進行PE75測序����,結果觀察到在ALK融合陽性樣本中,ALK的覆蓋表現出明顯的不對稱性����。這種不對稱性在樣本的外顯子20-24中尤為突出��,意味著這些區(qū)域的表達水平顯著高于其他外顯子�。具體來說�,在樣本?ALK_9?中,文章觀察到明顯的覆蓋不對稱�,與融合陽性狀態(tài)相匹配。以上表明每個樣本的融合狀態(tài)都可以通過其外顯子覆蓋的差異來推測�����。
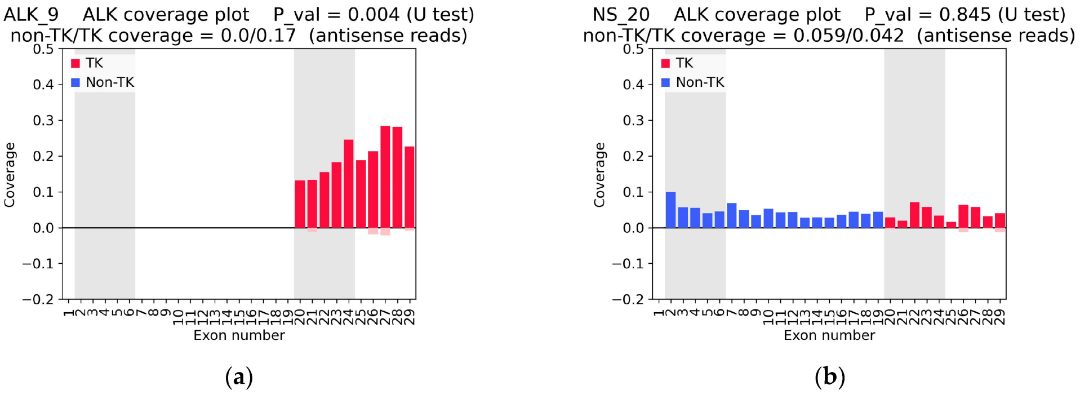
圖1?RNAseq數據的ALK覆蓋圖
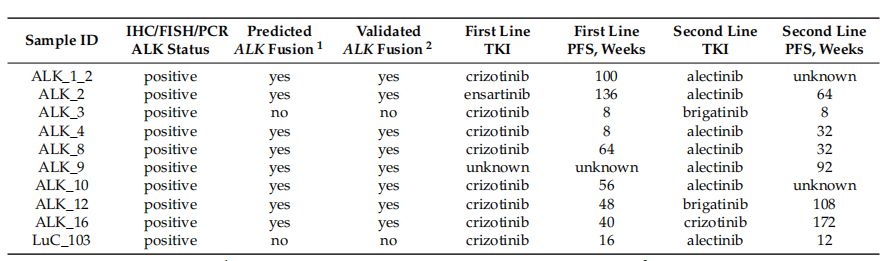
此外��,從表1中可以看到����,接受ALK靶向治療的患者中,預測為ALK融合陽性的組無進展生存期顯著延長����,進一步支持了文章方法的臨床相關性。表1 ALK靶向治療患者信息表
丨TruSight數據驗證ALK覆蓋不對稱
圖2展示了基于TruSight數據的ALK基因覆蓋圖�����,主要目的是分析不同樣本的ALK覆蓋不對稱性。數據顯示�����,確認有ALK融合的樣本在其ALK基因的覆蓋測量上表現出明顯的不對稱性(ALK_10)���,而在未確認融合的樣本中(NS_20)�����,這種不對稱性則不明顯。圖中的結果強調了ALK覆蓋不對稱性在腫瘤樣本中預測ALK融合的重要性�����,尤其是在基因的酪氨酸激酶(TK)區(qū)段的覆蓋情況���。
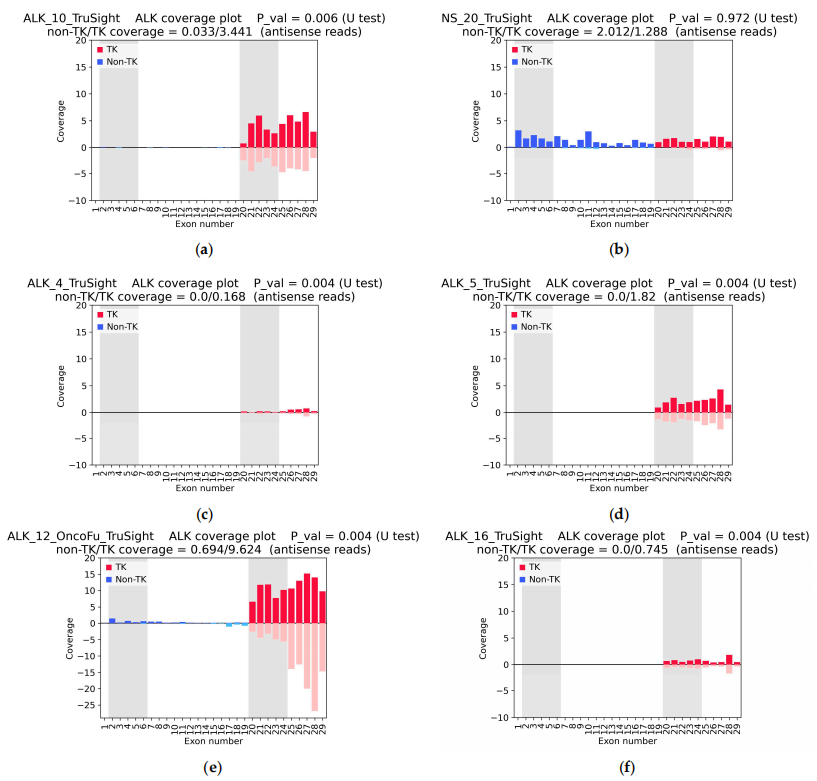
圖2?基于TruSight數據的ALK基因覆蓋結果圖
這項研究表明通過RNA測序外顯子覆蓋率分析����,能夠有效檢測ALK基因融合�。研究顯示,該新穎的生物信息學方法改善了傳統(tǒng)基因融合檢測技術的準確性����,能夠達到96%的驗證準確率�����,其中敏感性為100%���,特異性為94.9%。
Zakharova G, Suntsova M, Rabushko E, Mohammad T, Drobyshev A, Seryakov A, Poddubskaya E, Moisseev A, Smirnova A, Sorokin M, Tkachev V, Simonov A, Guguchkin E, Karpulevich E, Buzdin A. A New Approach of Detecting ALK Fusion Oncogenes by RNA Sequencing Exon Coverage Analysis. Cancers (Basel). 2024 Nov 16;16(22):3851. doi: 10.3390/cancers16223851. PMID: 39594806.