
9月15日��,真邁生物與序禎達(dá)生物��、UST共同簽署協(xié)議�����,正式達(dá)成戰(zhàn)略合作�。根據(jù)協(xié)議,序禎達(dá)生物將采用UST的TELL-Seq關(guān)聯(lián)長讀長二代測序技術(shù)�,并通過真邁生物的GenoLab M高通量基因測序平臺,推出經(jīng)濟(jì)可及的長讀長測序解決方案�����,為廣大合作伙伴提供高品質(zhì)的檢測分析服務(wù)�。作為此次戰(zhàn)略合作的第一個重量級成果,三方聯(lián)合推出的「細(xì)菌基因組完成圖-NGS躍級版」今日發(fā)布��。
它的詳情��,來一睹為快↓↓↓
細(xì)菌基因組完成圖-NGS躍級版
組裝細(xì)菌完整基因組是現(xiàn)代微生物研究的重要手段。利用細(xì)菌基因組完成圖可以更好地研究重要基因功能��、鑒定未知功能基因以及比較物種間的進(jìn)化關(guān)系����,這在傳染性疾病的研究�����、預(yù)防與治療�,環(huán)境微生物研究及疫苗和抗生物等的研究開發(fā)中都發(fā)揮著重要作用。
序禎達(dá)生物采用UST的TELL-Seq關(guān)聯(lián)長讀長二代測序技術(shù)�����,結(jié)合真邁生物的GenoLab M高通量基因測序平臺���,打造出一款通過二代測序平臺即能接近三代組裝效果的全新產(chǎn)品——細(xì)菌基因組完成圖-NGS躍級版����。
丨長讀長二代測序技術(shù)讓細(xì)菌基因組完成圖繪制觸手可及
細(xì)菌基因組完成圖-NGS躍級版采用了關(guān)聯(lián)長讀長二代測序技術(shù)——TELL-Seq����,該技術(shù)通過轉(zhuǎn)座酶對來自同一條長DNA的小片段進(jìn)行特定標(biāo)記,利用標(biāo)記對短讀長序列進(jìn)行拼接,讓二代測序的短讀長數(shù)據(jù)產(chǎn)生超長讀長測序效果���,等價讀長達(dá)20 kb-200 kb����。經(jīng)過創(chuàng)新開發(fā)的生信算法�,組裝的平均效果為contig(>10kb):1-10個;N50:2-5Mb(與基因組大小關(guān)聯(lián))�����。
該技術(shù)不僅可用于微生物de novo測序(菌種鑒定)��,還可應(yīng)用于基因組單倍體分型(Phasing)���、基因組結(jié)構(gòu)變異���、新物種全基因組de novo組裝等多個方向。

丨細(xì)菌基因組完成圖-NGS躍級版���,揭示細(xì)菌基因組完整面貌
細(xì)菌基因組完成圖-NGS躍級版通過基于TELL-Seq的二代測序技術(shù)以真邁生物的GenoLab M為載體����,對細(xì)菌基因組進(jìn)行測序,實(shí)現(xiàn)高性價比的基因組完成圖組裝���,獲得高準(zhǔn)確度的基因組信息���。

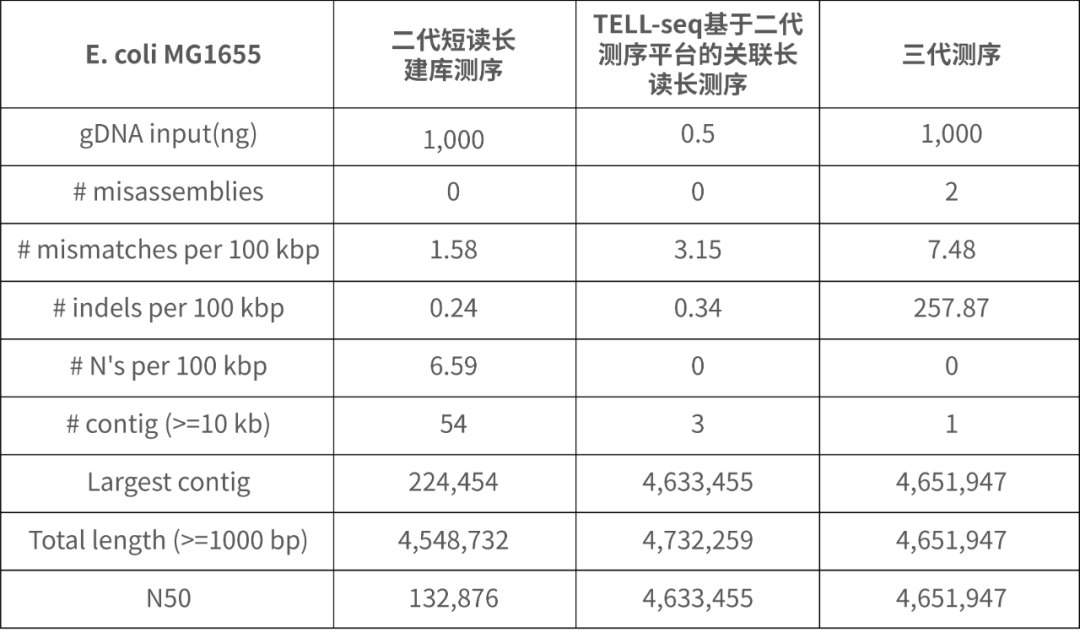
丨卓越效果,眼見為實(shí)
TELL-Seq技術(shù)的測序數(shù)據(jù)可以組裝出接近完美的細(xì)菌基因組
樣本要求及檢測周期:
送樣要求:≥50ng,片段長度>20kb��,未經(jīng)反復(fù)凍融���,無降解��,無污染
運(yùn)輸要求:-80℃保存�����,干冰運(yùn)輸
檢測周期:最短3周(收到樣本至交付組裝結(jié)果)
交付結(jié)果:DNA質(zhì)檢報告�����、測序下機(jī)原始數(shù)據(jù)�����、組裝結(jié)果
關(guān)于GenoLab M
GenoLab M是一款精準(zhǔn)�����、高效�、靈活、開放的桌面型高通量基因測序平臺�����,其采用基于芯片擴(kuò)增的表面熒光測序技術(shù)SURFSeq對堿基的熒光信號進(jìn)行識別���,實(shí)現(xiàn)邊合成邊測序�。GenoLab M可以兼容目前市面上主流的NGS文庫制備產(chǎn)品和數(shù)據(jù)分析流程。
相較于目前市場上的同類競品���,GenoLab M在擁有雙芯片平臺的基礎(chǔ)上���,還支持'滾動上機(jī)'模式,“滾動上機(jī)”是指GenoLab M在運(yùn)行單側(cè)芯片平臺測序時����,用戶可根據(jù)實(shí)際實(shí)驗(yàn)需求隨時啟動空閑芯片平臺的測試工作����,“雙芯片平臺+滾動上機(jī)模式”極大地提高了GenoLab M的檢測靈活性,為用戶帶來了優(yōu)秀的使用體驗(yàn)����。

參考文獻(xiàn):
1. Chen Z, Pham L, Wu TC, et al. Ultra-low input single tube linked-read library method enables short-read second-generation sequencing systems to generate highly accurate and economical long-range sequencing information routinely. Genome Res. 2020;30(6):898-909. doi: 10.1101/gr.260380.119.
2. Arun Sethuraman, Rosalina Stancheva, Ciara Sanders, et al. Genome of a novel Sediminibacterium discovered in association with two species of freshwater cyanobacteria from streams in Southern California, G3 Genes|Genomes|Genetics, Volume 12, Issue 7, July 2022, jkac123, https://doi.org/10.1093/g3journal/jkac123
3. Kumar, A., Im, K., Banjevic, M. et al. Whole-genome risk prediction of common diseases in human preimplantation embryos. Nat Med 28, 513–516 (2022). https://doi.org/10.1038/s41591-022-01735-0
4. Faith M Anderson, Noelle Visser, Kevin Amses, et al. Human commensal Candida albicans strains demonstrate substantial within-host diversity and retained pathogenic potential. bioRxiv 2022.09.09.507247; https://doi.org/10.1101/2022.09.09.507247