通過快速測序獲得新發(fā)�����、突發(fā)傳染病病原體的高質(zhì)量基因組序列對病原體致病機制研究、疾病溯源、疾病控制至關(guān)重要�����。FASTASeq 300支持自定義多個數(shù)據(jù)輸出的節(jié)點且最快可在19.5小時內(nèi)完成病原體基因組PE150測序,以更好地滿足用戶對檢測時效性和研究信息全面性的雙重需求�����。
為了評估FASTASeq 300在微生物de?novo組裝應(yīng)用上的表現(xiàn)�����,本次測評選擇了臨床常見致病菌——嗜肺性軍團菌1作為測試樣本構(gòu)建文庫�����,文庫分別在FASTASeq 300���、GenoLab M和NV測序平臺進行PE150測序�����,均取100×數(shù)據(jù)進行分析�����。測評結(jié)果顯示�,F(xiàn)ASTASeq 300測序數(shù)據(jù)質(zhì)量高、序列組裝結(jié)果與參考基因組具有較高一致性���,其進化分析與參考基因組能準確聚類于同一分支���。
測序數(shù)據(jù)高
FASTASeq 300測序數(shù)據(jù)Raw data Q20大于97%,Q30大于92%�,Clean reads/Raw reads比值平均大于0.99,測序質(zhì)量表現(xiàn)優(yōu)異��。

圖1. FASTASeq 300�、NV和GenoLab M平臺測序質(zhì)量表現(xiàn)
組裝質(zhì)量好
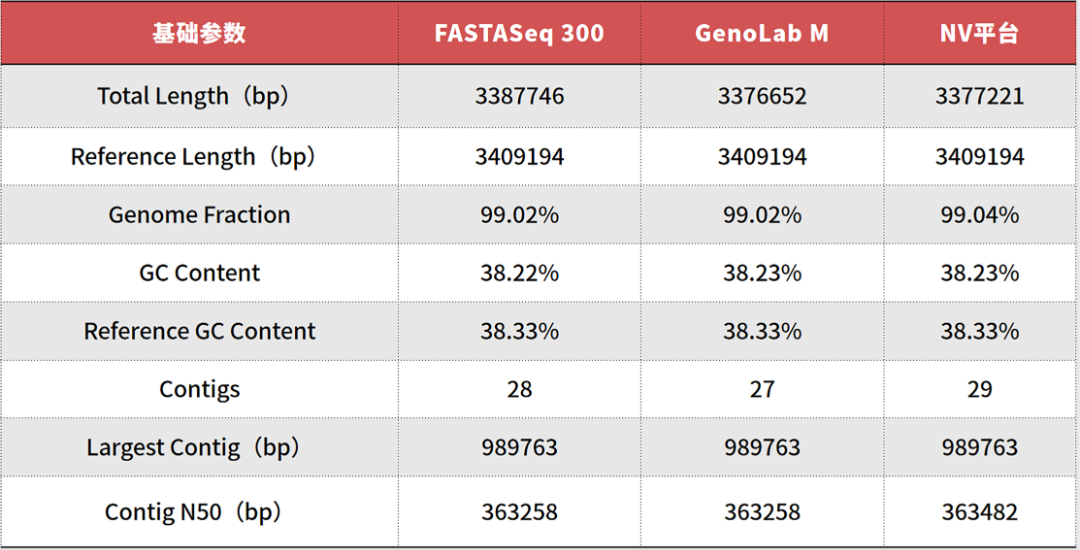
與參考基因組序列進行比對分析,結(jié)果表明FASTASeq 300測序數(shù)據(jù)在組裝質(zhì)量方面與對比平臺表現(xiàn)一致�,Largest?Contig接近基因組全長的1/3。
表1. 組裝結(jié)果評價指標
組裝完整性高
BUSCO2和Circos3圖展示了FASTASeq 300測序數(shù)據(jù)組裝結(jié)果的高完整性和準確性�。
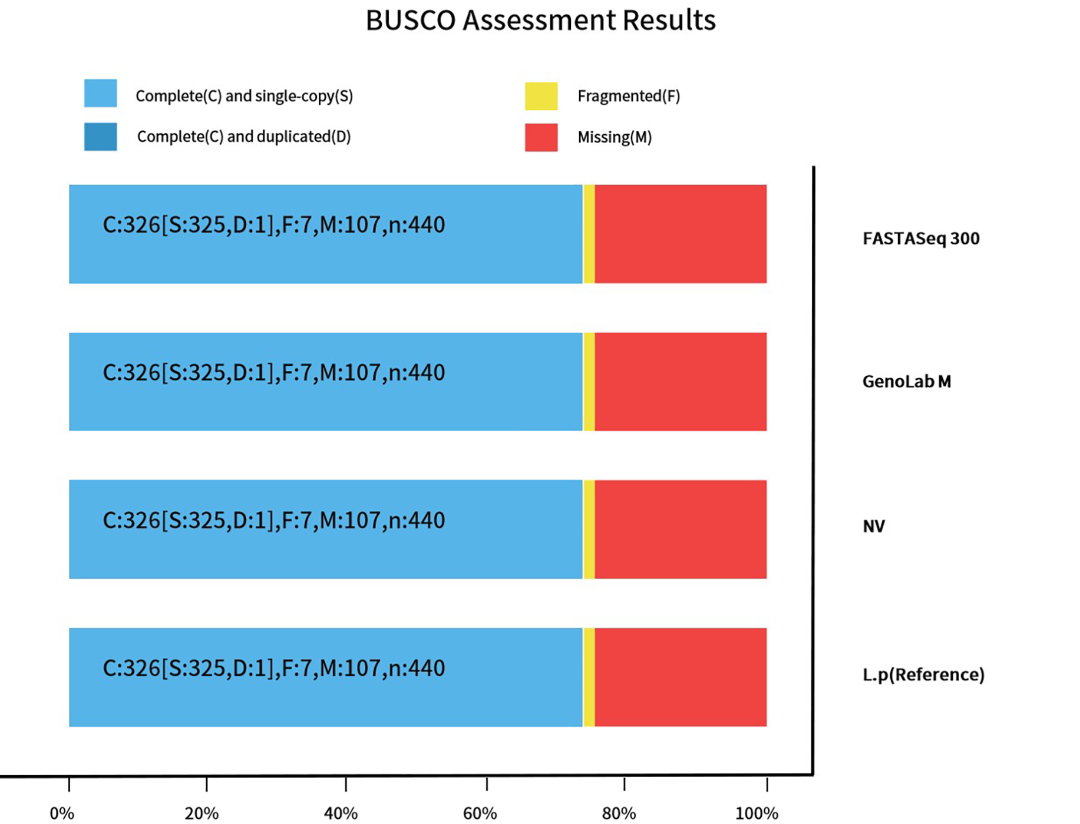
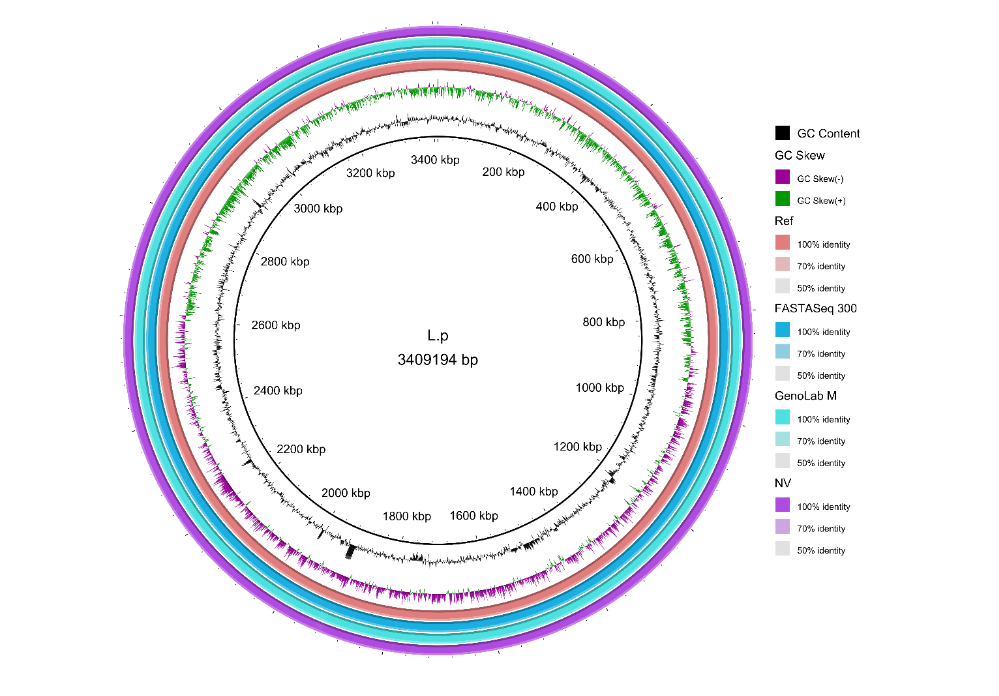
圖2. ?BUSCO 組裝結(jié)果和Circos 基因組完整性分析結(jié)果圖
進化分析
基于FASTASeq 300測序數(shù)據(jù)組裝的基因組序列及其22同種不同株的基因組序列構(gòu)建系統(tǒng)進化樹�,結(jié)果顯示FASTASeq 300測序數(shù)據(jù)組裝的基因組序列結(jié)果與參考基因組能準確地聚類到進化樹同一分支�。

FASTASeq 300在微生物基因組de?novo組裝應(yīng)用方向上性能優(yōu)異,其快速��、靈活�����、準確的特性可很好地滿足病原體全基因組測序的要求�。未來FASTASeq 300將助力更多微生物組學領(lǐng)域的科學研究和臨床應(yīng)用。
更多精彩測評����,敬請期待�����!

1. 嗜肺性軍團菌(Legionella pneumophila):全基因組大小約3.4 Mb��,GC含量38.3%��,是一種廣泛存在于自然界中的機會致病菌��,能引起以發(fā)熱和呼吸道癥狀為主的疾病�。
2. BUSCO組裝評價:使用相近物種之間的一些保守序列構(gòu)建單拷貝基因集數(shù)據(jù)庫���,與組裝結(jié)果進行比對,從組裝結(jié)果比對上的比例��、完整性����,來評價組裝結(jié)果的準確性和完整性。
3. Circos組裝評價:采用Circos軟件對測序結(jié)果進行分析����,通過組裝圖譜的形式直觀地表現(xiàn)出組裝數(shù)據(jù)對參考基因組覆蓋程度。